Editing genetico
Da tempo si usano le tecnologie del DNA ricombinante per modificare il genoma di diversi organismi, ma le modifiche non sono sempre precise: il DNA esogeno si colloca spesso in punti casuali e non sostituisce quello alterato ma semplicemente lo affianca. L'editing genetico consiste nell'eliminare, sostituire o inserire del DNA in siti specifici con una precisione assai superiore rispetto alle tecniche di ingegneria genetica precedentemente adoperate.
Per l'editing si impiegano delle nucleasi come forbici molecolari in grado di tagliare il DNA in siti specifici. Quando è avvenuto il taglio, la cellula attiva i sistemi di autoriparazione per ricongiungere i monconi. Se, oltre alla nucleasi, si fornisce anche una copia del gene di interesse che funzioni da stampo, questa potrà guidare la riparazione attraverso il meccanismo della ricombinazione omologa. Si può anche fornire il DNA che guida la riparazione, contenente un nuovo gene e così si ottiene un organismo transgenico.
Tra i metodi attualmente a disposizione per il taglio del DNA citiamo le TALEN e il sistema CRISPR/Cas9.
TALEN
Le endonucleasi (EN) TAL → TALEN (Transcription Activator-Like Effector Nucleases cioè effettori simili agli attivatori della trascrizione), sono delle proteine modulari di circa 34 amminoacidi, ripetuti molte volte, che Batteri del genere Xanthomonas inseriscono nelle piante. Questi effettori, quando giungono al nucleo, attivano dei geni che rendono la pianta suscettibile alle infezioni.

Effettore TAL che avvolge il DNA
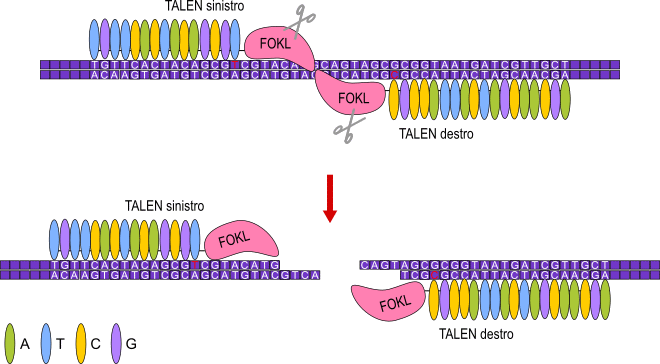
Le TALEN possono essere create artificialmente per l'editing genetico unendo due nucleasi chiamate Fokl e proteine ricavate dai fattori di trascrizione dei Batteri. Ogni monomero della proteina può riconoscere un solo nucleotide quando l'effettore TAL si lega al DNA perciò, unendo più monomeri, si può preparare un dominio che riconosca una specifica sequenza di DNA della cellula bersaglio. A questo punto la nucleasi taglia entrambe le catene del DNA e quindi può essere usata per bloccare un gene specifico o per inserire un nuovo gene ingegnerizzato sfruttando il naturale meccanismo di riparazione del DNA della cellula.
CRISPR/Cas9
Fagi e plasmidi quando infettano una cellula procariote inseriscono dei segmenti di DNA chiamati CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats, cioè brevi ripetizioni palindrome raggruppate e separate a intervalli regolari [pronuncia crìsper]). Si è scoperto che i Batteri possiedono la proteina Cas9 che cerca le CRISPR, taglia il segmento funzionando come una forbice molecolare, e rimuove il DNA virale che si è inserito nel cromosoma della cellula. È una specie di difesa immunitaria dei Procarioti che rimane memorizzata per futuri attacchi ed è trasmessa alle generazioni successive. Questa capacità la possiamo sfruttare programmando la Cas9 per tagliare sequenze specifiche di DNA negli animali, nelle piante e nell'uomo in modo efficace ed economico, onde eliminare interi geni, correggere geni mutati, silenziare geni difettosi o inserire un segmento di DNA esogeno.
Per indirizzare la Cas9 verso la sequenza bersaglio che si vuole modificare, si costruisce un RNA guida, complementare alla sequenza del DNA bersaglio, che si associa alla proteina. L'RNA si ancora al bersaglio, la Cas9 taglia il DNA, elimina, sostituisce o inserisce nuove sequenze e ricuce la doppia elica.
Questo sistema offre una possibilità nuova per la cura di diverse malattie genetiche. Purtroppo, il taglio del genoma umano non si è dimostrato sempre preciso perché si sono avuti degli errori di taglio (off-target) che ne hanno frenato l'impiego. Le maggiori obiezioni sono però di carattere etico, soprattutto quando riguardano l'editing degli embrioni umani, che può essere fatto per eliminare un difetto genetico, ma anche per favorire particolari caratteristiche, le cui modifiche saranno trasmesse alla prole.

La genetica inversa
La genetica classica, davanti a un particolare fenotipo, cerca di individuare le basi genetiche che lo producono. Al contrario, con la genetica inversa, avendo a disposizione una sequenza genica, si cerca di scoprirne la funzione a livello di fenotipo.

Il sequenziamento automatico dei genomi ha prodotto una grande quantità di sequenze geniche, molte delle quali hanno una funzione ancora sconosciuta, che sono disponibili online. I ricercatori devono capire qual è la loro funzione e per fare questo, con tecniche diverse, ne mutano la sequenza per vedere gli effetti fenotipici sulle cellule o su modelli animali.
Oltre agli agenti mutageni usati nella genetica classica: agenti chimici, radiazioni o elementi trasponibili, che inducono mutazioni casuali nel genoma, oggi i ricercatori hanno a disposizione nuovi strumenti tecnologici, di cui abbiamo parlato in precedenza (knockout genico, Rna interference) che consentono mutazioni gene-specifiche senza avere la necessità di controllare tutto il genoma.
La genomica
La genomica è quel campo della biologia molecolare che studia la struttura, l'informazione, la funzione e l'evoluzione del genoma di una particolare specie.
La genomica è a sua volta suddivisa in sottosezioni.
La genomica strutturale si occupa del sequenziamento, quantificazione e mappatura genetica (quali geni sono presenti) e fisica (disposizione dei geni) di un genoma.
La genomica funzionale studia le funzioni dei geni, le reciproche interazioni, le relazioni con i loro prodotti e come dirigono lo sviluppo di un organismo. Il genoma non comprende solo geni codificanti per le proteine che, come abbiamo visto, sono una minoranza, ma anche geni di regolazione, insieme a moltissime sequenze la cui funzione non è ancora conosciuta. Per connotarli funzionalmente si ricorre alla comparazione con altri geni, di cui si conosce la funzione, presenti all'interno di banche dati, accessibili tramite internet.
Un'altra modalità per scoprire la funzione di un gene è disattivarlo con knock-out genico o RNA interference per vedere gli effetti sul fenotipo.
La conoscenza dell'intero genoma è di grande utilità in campo biomedico. Molte malattie, infatti, sono multifattoriali e la visione complessiva del genoma permette di individuare più facilmente i geni coinvolti e come interagiscono tra loro.
Per la grande quantità di dati biologici ricavati in laboratorio, questa disciplina si basa sulla bioinformatica (vedi sotto).
La genomica comparata o comparativa confronta i genomi di diverse specie relativamente alle sequenze a alla loro organizzazione. La comparazione consente di distinguere a livello molecolare una specie dall'altra e di individuare i geni presenti in una specie e assenti in un'altra, onde poterne correlare la relativa fisiologia, nonché per stabilire le parentele evolutive.
Il principio su cui si basa la comparazione è che tutti gli organismi attuali derivino da un antenato comune. Con il trascorrere del tempo, si accumulano mutazioni nel genoma: in base al numero di differenze genetiche tra due specie è possibile ricavare la loro distanza evolutiva e il momento in cui si sono separate da un progenitore comune.
Il confronto tra le specie ci mostra anche la presenza di geni che si sono conservati lungo il percorso evolutivo perché svolgono funzioni fondamentali per la vita e i geni nuovi, unici per ogni specie, selezionati per svolgere particolari funzioni.
La metagenomica studia, mediante le moderne tecniche genomiche, le comunità microbiche direttamente nel loro ambiente naturale, per valutarne la biodiversità genetica. Il metagenoma, cioè tutte le sequenze degli organismi presenti nell'ambiente in esame, consente sia di identificare le specie presenti che, soprattutto, di cercare geni di interesse per un determinato scopo.

Genoma mitocondriale umano
La trascrittomica
La trascrittomica è la disciplina che studia il trascrittoma, cioè l'insieme completo dei trascritti di RNA messaggero di una cellula o di un tessuto. Si differenzia dal proteoma (vedi sotto) perché solo una parte degli mRNA si esprime in proteine. Il trascrittoma è molto dinamico perché varia in base agli stimoli ricevuti in un determinato momento e allo stadio di vita e di condizioni della cellula e si traduce nel fenotipo.
Lo scopo della trascrittomica è classificare l'espressione di tutti i trascritti e a quantificare le variazioni di espressione di ogni trascrizione utilizzando le tecniche di microarray di DNA e dell'RNA-seq.
La trascrittomica trova ampia applicazione nel campo medico per la prognosi e la diagnosi precoci di malattie e per lo sviluppo di terapie mirate.
Struttura cristallina dell'enzima PNPasi che demolisce la catena di RNA
La proteomica
Si chiama proteoma (termine che deriva dalla fusione di proteine + genoma) l'insieme delle proteine effettivamente espresse in una cellula o in un tessuto in un dato momento, comprese le isoforme e le modificazioni post-traduzionali.
La proteomica è quella branca della biologia molecolare che studia le proteine presenti in un campione dal punto di vista strutturale, funzionale e di interazione. Mentre il genoma rimane costante nel tempo e in tutte le cellule dell'individuo, le proteine espresse si modificano in risposta a stimoli esterni e in base al tipo di tessuto e al ciclo di vita, perciò ad un genoma corrispondono più proteomi. Il numero delle proteine espresse varia da centomila al milione eppure i geni codificanti sono solo 20000. Questa discrepanza si spiega perché esistono promotori e terminatori alternativi per ciascun gene, splicing alternativo dei trascritti e modifiche post-traduzionali. A un gene, quindi, possono corrispondere più proteine.
La proteomica viene suddivisa in tre grandi settori.
La proteomica di espressione riguarda la localizzazione, la qualità e la quantità di prodotti espressi e la loro differenziazione in determinate condizioni quali la crescita, il differenziamento, l'invecchiamento e la malattia.
La proteomica strutturale (o sistematica) identifica e caratterizza le proteine in riferimento alla struttura tridimensionale.
La proteomica funzionale analizza le interazioni che le proteine stabilisco con altre proteine a seconda delle condizioni indicate sopra (interattomica). Si occupa anche delle modificazioni post-traduzionali e delle interazioni tra una proteina e il suo substrato, cioè i processi chimici che coinvolgono i metaboliti (metabolomica), nonché le funzioni specifiche di ciascuna.
Per studiare il proteoma si parte dall'estrazione delle proteine dalla cellula - lisata meccanicamente, con ultrasuoni o detergenti - tramite centrifugazione. Per l'analisi dell'estratto ci si avvale della spettrometria di massa, l'elettroforesi, la cromatografia o microarray. Sono disponibili anche banche dati contenenti tutte le possibili sequenze geniche espresse, in modo da correlare la proteina individuata con il gene corrispondente e software per l'analisi dei dati.
Le applicazioni della proteomica sono molteplici. Con l'ingegneria metabolica si possono alterare le vie metaboliche per migliorare prodotti, aumentarne la quantità o far loro acquisire caratteristiche che prima non possedevano (ad es. il pomodoro bronzeo ricco di polifenoli).
Conoscendo la struttura tridimensionale di una proteina, si possono progettare farmaci mirati, più efficaci e tollerabili.
Poiché una patologia è spesso accompagnata dalla produzione di proteine specifiche o alterate, si possono identificare nuovi marcatori per una diagnosi precoce e una terapia mirata, comprendere le fasi e i meccanismi di sviluppo delle malattie tumorali.

Analizzatore di pattern proteici
La bioinformatica
La bioinformatica è quella che abbiamo definito biotecnologia oro. I dati forniti dalla genomica e dalla proteomica devono essere analizzati elaborati e archiviati per essere operativi. La bioinformatica applica i metodi matematici, statistici e computazionali per lo studio e l'interpretazione dei dati biologici a livello molecolare. Grazie al computer si possono raccogliere, analizzare e gestire le informazioni genetiche e biochimiche che, con le nuove tecnologie, arrivano in grande quantità, non più analizzabili a mano, e sviluppare strumenti per le nuove esigenze.
Da questi dati si possono ricavare modelli biologici interpretativi per correlare la struttura genetica ai processi metabolici e alla fisiologia degli organismi. Si posso progettare software per ricostruire la forma tridimensionale delle proteine a partire dalla sequenza del DNA, per identificare i gruppi funzionali o anche per modificarle. Il sequenziamento dei genomi sarebbe impossibile senza computer e software dedicati. Anche l'analisi delle impronte genetiche, sempre più richieste, sarebbe assai lenta.
L'identificazione delle sequenze del genoma è solo un primo passo. Sono ancora sconosciute molte funzioni biologiche corrispondenti alle sequenze nucleotidiche, perché solo alcune codificano per le proteine, mentre altre sono segnali, meccanismi di riconoscimento o molte restano da identificare. Per questo, quando si individua una nuova sequenza la si confronta con quelle presenti nelle grandi banche dati mondiali per cercare somiglianze statisticamente significative. Queste banche dati nascono proprio per la necessità di immagazzinare, catalogare, facilitare l'interrogazione e analizzare l'enorme mole di dati e ciò sarebbe stato impossibile senza le nuove tecnologie informatiche e telematiche e lo sviluppo di algoritmi, strumenti di statistica e software in grado di verificare un gran numero di relazioni.
La bioinformatica assume grande importanza nella ricerca medica sia studiando le relazioni tra una patologia e la componente genetica e ambientale che per lo sviluppo di farmaci personalizzati. Si è osservato, infatti, che un farmaco agisce in modo diverso nelle varie persone e questo per la variabilità dovuta agli SNP.
La biologia evolutiva, grazie alla possibilità di confrontare i genomi di diversi organismi, ha consentito di ricostruire in modo più preciso l'albero filogenetico.


